Pseudogen: Perbedaan antara revisi
Tidak ada ringkasan suntingan Tag: Suntingan visualeditor-wikitext |
Tidak ada ringkasan suntingan Tag: Suntingan visualeditor-wikitext |
||
| Baris 1: | Baris 1: | ||
[[File:Pseudogene defects.png|alt=Drawing of a gene showing kinds of defects (missing promoter, start codon or introns, premature stop codon, frameshift mutation, partial deletion).|540x540px|thumb]] |
[[File:Pseudogene defects.png|alt=Drawing of a gene showing kinds of defects (missing promoter, start codon or introns, premature stop codon, frameshift mutation, partial deletion).|540x540px|thumb]] |
||
'''Pseudogen''' ([[Bahasa Inggris|Inggris]]; ''Pseudogenes'') adalah salinan [[Gen|gen]] yang biasanya tidak memiliki [[intron]] dan urutan [[DNA]], bisa juga diartikan sebagai [[segmen]] |
'''Pseudogen''' ([[Bahasa Inggris|Inggris]]; ''Pseudogenes'') adalah salinan [[Gen|gen]] yang biasanya tidak memiliki [[intron]] dan urutan [[DNA]], dan bisa juga diartikan sebagai [[segmen]] non-fungsional dari [[DNA]] yang menyerupai fungsional asli dari [[Gen|gen]] itu sendiri.<ref name="PSEUDOGEN">{{cite web|url=https://www.institutoroche.es/recursos/glosario/pseudogen|title=Pseudogen|website=www.institutoroche.es|accessdate=22 November 2020|lang=es}}</ref> Meskipun pseudogen secara genetik terlihat mirip dengan gen fungsional asli, namun pseudogen sudah mengalami banyak [[mutasi]]. Sebagian besar pseudogen muncul sebagai salinan gen fungsional yang berlebihan, baik secara langsung oleh [[duplikasi]] DNA ataupun juga secara tidak langsung oleh [[Transkripsi balik|transkripsi]] dari transkrip [[mRNA]]. Dan ini biasanya akan teridentifikasi ketika dilakukan analisis urutan [[genom]] dan urutan yang mirip gen namun tidak memiliki urutan pengaturan yang diperlukan untuk dilakukannya [[Transkripsi (genetik)|transkripsi]] atau [[Terjemahan (biologi)|terjemahan]]. Urutan pengkodeannya rusak karena [[Mutasi frameshift|frameshift]] atau prematur [[Stop kodon]]. |
||
Kebanyakan genom non-bakteri mengandung banyak pseudogen, |
Kebanyakan [[genom]] non-bakteri mengandung banyak pseudogen, dan bisa sebanyak gen fungsional. Ini terjadi karena berbagai proses biologis secara tidak sengaja membuat pseudogen, dan tidak ada mekanisme khusus untuk menghilangkannya dari [[genom]]. Pada akhirnya pseudogen dapat dihapus dari genomnya secara kebetulan melalui [[replikasi DNA]] atau [[perbaikan DNA]], atau bisa juga pseudogen dapat mengakumulasi banyak perubahan [[Mutasi|mutasi]] sehingga tidak lagi dapat dikenali sebagai gen sebelumnya. Analisis peristiwa degenerasi ini membantu memperjelas efek proses non-selektif pada [[genom]]. Sekuens pseudogen dapat ditranskripsi menjadi [[RNA]] pada tingkat rendah, karena elemen [[Promotor (genetika)|promotor]] diwarisi dari gen leluhur atau timbul oleh mutasi baru. |
||
Sekuens pseudogen dapat ditranskripsi menjadi [[RNA]] pada tingkat rendah, karena elemen [[Promotor (genetika) | promotor]] diwarisi dari gen leluhur atau timbul oleh mutasi baru. Meskipun sebagian besar transkrip ini tidak memiliki signifikansi fungsional yang lebih daripada transkrip kebetulan dari bagian lain genom, beberapa telah menghasilkan RNA pengaturan yang bermanfaat dan protein baru.. |
|||
==Tipe-tipe dan Keaslian == |
==Tipe-tipe dan Keaslian == |
||
| Baris 15: | Baris 13: | ||
[[File:Pseudogene2jpg.jpg|thumb|Produksi pseudogen yang diproses|alt=|330x330px]] |
[[File:Pseudogene2jpg.jpg|thumb|Produksi pseudogen yang diproses|alt=|330x330px]] |
||
Pada [[eukariota]] yang lebih tinggi, secara khusus [[mamalia]], [[retrotransposisi]] merupakan peristiwa yang lumrah terjadi dan memiliki pengaruh besar pada komposisi [[genom]]. Misalnya, |
Pada [[eukariota]] yang lebih tinggi, secara khusus [[mamalia]], [[Retrotransposisi|retrotransposisi]] merupakan peristiwa yang lumrah terjadi dan memiliki pengaruh besar pada komposisi [[genom]]. Misalnya, sekitar 30-44% [[genom manusia]] terdiri dari elemen berulang seperti [[Retrotransposon#SINEs|SINEs]] dan [[Retrotransposon#LINEs|LINEs]].<ref name="Jurka_2004">{{cite journal |vauthors= Jurka J |title= Evolutionary impact of human Alu repetitive elements |journal= Current Opinion in Genetics & Development |volume= 14 |issue= 6 |pages= 603–8 |date= December 2004 |pmid= 15531153 |doi= 10.1016/j.gde.2004.08.008}}</ref><ref name="Dewannieux_2005">{{cite journal |vauthors= Dewannieux M, Heidmann T |title= LINEs, SINEs and processed pseudogenes: parasitic strategies for genome modeling |journal= Cytogenetic and Genome Research |volume= 110 |issue= 1–4 |pages= 35–48 |year= 2005 |pmid= 16093656 |doi= 10.1159/000084936|s2cid= 25083962 }}</ref> Di dalam proses terjadinya retrotransposisi ini, sebagian dari transkrip [[Gen|gen]] [[mRNA|messenger RNA]] (mRNA) atau [[hnRNA]] secara otomatis terjadi [[Transkripsi balik|transkripsi balik]] dan akan kembali ke [[DNA]] dan dimasukkan ke dalam [[kromosom]] DNA. Meskipun retrotransposon membuat salinan dirinya sendiri, sistem '' in vitro '' menunjukkan bahwa mereka juga dapat membuat salinan gen secara acak yang dapat ditransposisikan kembali.<ref name="Dewannieux_2003">{{cite journal |vauthors= Dewannieux M, Esnault C, Heidmann T |title= LINE-mediated retrotransposition of marked Alu sequences |journal= Nature Genetics |volume= 35 |issue= 1 |pages= 41–8 |date= September 2003 |pmid= 12897783 |doi= 10.1038/ng1223|s2cid= 32151696 }}</ref> |
||
Setelah pseudogen ini dimasukkan kembali ke dalam genom, pseudogen tersebut |
Setelah pseudogen ini dimasukkan kembali ke dalam [[genom]], pseudogen tersebut akan mengandung [[poliadenilasi]] (ekor poly-A), dan biasanya akan memiliki [[intron]] yang disambung, dan keduanya merupakan ciri khas [[cDNA]]. Namun, karena berasal dari produk RNA, pseudogen yang diproses akan kekurangan [[promotor|promotor hulu]] dari gen normal, maka mereka dianggap "mati pada saat kedatangan", segera menjadi pseudogen non-fungsional setelah dilakukan peristiwa retrotransposisi.<ref name="Graur_1989">{{cite journal |vauthors= Graur D, Shuali Y, Li WH |title= Deletions in processed pseudogenes accumulate faster in rodents than in humans |journal= Journal of Molecular Evolution |volume= 28 |issue= 4 |pages= 279–85 |date= April 1989 |pmid= 2499684 |doi= 10.1007/BF02103423|bibcode= 1989JMolE..28..279G |s2cid= 22437436 }}</ref> |
||
Penyisipan ini terkadang menyumbangkan [[ekson]] ke gen yang ada, biasanya melalui transkrip [[Splicing alternatif|penyambungan alternatif]],<ref name="Baertsch_2008">{{cite journal |vauthors= Baertsch R, Diekhans M, Kent WJ, Haussler D, Brosius J |title= Retrocopy contributions to the evolution of the human genome |journal= BMC Genomics |volume= 9 |pages= 466 |date= October 2008 |pmid= 18842134 |pmc= 2584115 |doi= 10.1186/1471-2164-9-466}}</ref><ref name="Pavlicek_2002">{{cite journal |vauthors= Pavlícek A, Paces J, Zíka R, Hejnar J |title= Length distribution of long interspersed nucleotide elements (LINEs) and processed pseudogenes of human endogenous retroviruses: implications for retrotransposition and pseudogene detection |journal= Gene |volume= 300 |issue= 1–2 |pages= 189–94 |date= October 2002 |pmid= 12468100 |doi= 10.1016/S0378-1119(02)01047-8}}</ref> dan pseudogen yang diproses secara terus menerus akan dibuat pada [[primata]].<ref>{{cite journal |vauthors= Navarro FC, Galante PA |title= A Genome-Wide Landscape of Retrocopies in Primate Genomes |journal= Genome Biology and Evolution |volume= 7 |issue= 8 |pages= 2265–75 |date= July 2015 |pmid= 26224704 |pmc= 4558860 |doi= 10.1093/gbe/evv142}}</ref> Contoh yang bisa diambil adalah populasi manusia, yang memiliki kumpulan pseudogen olahan yang berbeda dari setiap individu.<ref>{{cite journal |vauthors= Schrider DR, Navarro FC, Galante PA, Parmigiani RB, Camargo AA, Hahn MW, de Souza SJ |title= Gene copy-number polymorphism caused by retrotransposition in humans |journal= PLOS Genetics |volume= 9 |issue= 1 |pages= e1003242 |date= 2013-01-24 |pmid= 23359205 |pmc= 3554589 |doi= 10.1371/journal.pgen.1003242}}</ref> |
|||
===Tidak-diproses=== |
===Tidak-diproses=== |
||
Revisi per 23 November 2020 08.12

Pseudogen (Inggris; Pseudogenes) adalah salinan gen yang biasanya tidak memiliki intron dan urutan DNA, dan bisa juga diartikan sebagai segmen non-fungsional dari DNA yang menyerupai fungsional asli dari gen itu sendiri.[1] Meskipun pseudogen secara genetik terlihat mirip dengan gen fungsional asli, namun pseudogen sudah mengalami banyak mutasi. Sebagian besar pseudogen muncul sebagai salinan gen fungsional yang berlebihan, baik secara langsung oleh duplikasi DNA ataupun juga secara tidak langsung oleh transkripsi dari transkrip mRNA. Dan ini biasanya akan teridentifikasi ketika dilakukan analisis urutan genom dan urutan yang mirip gen namun tidak memiliki urutan pengaturan yang diperlukan untuk dilakukannya transkripsi atau terjemahan. Urutan pengkodeannya rusak karena frameshift atau prematur Stop kodon.
Kebanyakan genom non-bakteri mengandung banyak pseudogen, dan bisa sebanyak gen fungsional. Ini terjadi karena berbagai proses biologis secara tidak sengaja membuat pseudogen, dan tidak ada mekanisme khusus untuk menghilangkannya dari genom. Pada akhirnya pseudogen dapat dihapus dari genomnya secara kebetulan melalui replikasi DNA atau perbaikan DNA, atau bisa juga pseudogen dapat mengakumulasi banyak perubahan mutasi sehingga tidak lagi dapat dikenali sebagai gen sebelumnya. Analisis peristiwa degenerasi ini membantu memperjelas efek proses non-selektif pada genom. Sekuens pseudogen dapat ditranskripsi menjadi RNA pada tingkat rendah, karena elemen promotor diwarisi dari gen leluhur atau timbul oleh mutasi baru.
Tipe-tipe dan Keaslian
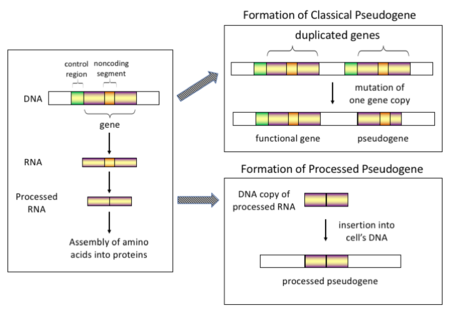
Ada empat jenis utama dalam pseudogen, dan keempat jenis ini memiliki mekanisme asal dan ciri khas yang berbeda. Empat jenis utama pseudogen tersebut ialah:
Diproses

Pada eukariota yang lebih tinggi, secara khusus mamalia, retrotransposisi merupakan peristiwa yang lumrah terjadi dan memiliki pengaruh besar pada komposisi genom. Misalnya, sekitar 30-44% genom manusia terdiri dari elemen berulang seperti SINEs dan LINEs.[4][5] Di dalam proses terjadinya retrotransposisi ini, sebagian dari transkrip gen messenger RNA (mRNA) atau hnRNA secara otomatis terjadi transkripsi balik dan akan kembali ke DNA dan dimasukkan ke dalam kromosom DNA. Meskipun retrotransposon membuat salinan dirinya sendiri, sistem in vitro menunjukkan bahwa mereka juga dapat membuat salinan gen secara acak yang dapat ditransposisikan kembali.[6]
Setelah pseudogen ini dimasukkan kembali ke dalam genom, pseudogen tersebut akan mengandung poliadenilasi (ekor poly-A), dan biasanya akan memiliki intron yang disambung, dan keduanya merupakan ciri khas cDNA. Namun, karena berasal dari produk RNA, pseudogen yang diproses akan kekurangan promotor hulu dari gen normal, maka mereka dianggap "mati pada saat kedatangan", segera menjadi pseudogen non-fungsional setelah dilakukan peristiwa retrotransposisi.[7]
Penyisipan ini terkadang menyumbangkan ekson ke gen yang ada, biasanya melalui transkrip penyambungan alternatif,[8][9] dan pseudogen yang diproses secara terus menerus akan dibuat pada primata.[10] Contoh yang bisa diambil adalah populasi manusia, yang memiliki kumpulan pseudogen olahan yang berbeda dari setiap individu.[11]
Tidak-diproses

Tidak-diproses (Inggris: Non-processed) disebut juga pseudogen duplikat. Duplikasi genetik adalah suatu proses yang umum dan penting dalam terjadinya evolusi genom. Salinan dari gen fungsional dapat muncul akibat dari peristiwa duplikasi gen yang disebabkan oleh rekombinasi homolog misalnya, sekuens berulang sine pada kromosom yang tidak sejajar dan kemudian memperoleh mutasi yang menyebabkan salinan kehilangan fungsi gen aslinya. Pseudogen duplikat biasanya memiliki semua karakteristik yang sama dengan gen, termasuk struktur ekson - intron utuh dan urutan pengaturan. Hilangnya fungsi gen duplikat biasanya memiliki pengaruh kecil pada kebugaran suatu organisme, karena salinan fungsional yang utuh masih ada. Menurut beberapa model evolusi, pseudogen duplikat yang dibagikan menunjukkan keterkaitan evolusioner manusia dengan primata lainnya.[12]
Jika pseudogenisasi disebabkan oleh duplikasi gen, biasanya terjadi dalam beberapa juta tahun pertama setelah duplikasi gen, asalkan gen tersebut tidak mengalami tekanan seleksi.[13] Duplikasi gen menghasilkan fungsional redundansi dan biasanya tidak menguntungkan untuk membawa dua gen identik. Mutasi yang mengganggu struktur atau fungsi salah satu dari kedua gen tidak merusak dan tidak akan dihilangkan melalui proses seleksi.
Akibatnya, gen yang telah bermutasi secara bertahap menjadi pseudogen dan akan menjadi tidak terekspresikan atau tidak berfungsi. Nasib evolusioner semacam ini ditunjukkan oleh populasi model genetik[14][15] dan juga oleh analisis genom.[13][16] Menurut konteks evolusi, pseudogen ini akan dihapus atau menjadi sangat berbeda dari gen induk sehingga tidak dapat diidentifikasi lagi. Pseudogen yang relatif muda dapat dikenali karena kemiripan urutannya.[17]
Pseudogen kesatuan

Berbagai mutasi (seperti indel dan mutasi nonsense) dapat mencegah gen menjadi normal ditranskripsikan atau diterjemahkan, dan sehingga gen tersebut menjadi kurang- atau tidak berfungsi atau "dinonaktifkan". Ini adalah mekanisme yang sama dimana gen yang tidak diproses menjadi pseudogen, tetapi perbedaan dalam kasus ini adalah bahwa gen tidak diduplikasi sebelum pseudogenisasi. Biasanya, pseudogen seperti itu tidak mungkin menjadi tetap dalam suatu populasi, tetapi berbagai efek populasi, seperti pergeseran genetik, hambatan populasi, atau, dalam beberapa kasus, seleksi alam, dapat menyebabkan fiksasi. Contoh klasik dari pseudogen kesatuan adalah gen yang diduga mengkodekan enzim L-gulono-γ-lactone oxidase (GULO) pada primata. Pada semua mamalia yang dipelajari selain primata (kecuali marmut), GULO membantu biosintesis asam askorbat (vitamin C), tetapi ia ada sebagai gen yang cacat (GULOP) pada manusia dan primata lainnya.[18][19]
Contoh lain yang lebih baru dari gen yang cacat menghubungkan deaktivasi gen caspase 12 (melalui mutasi nonsense) ke seleksi positif pada manusia.[20] Telah dibuktikan bahwa pseudogen yang diproses mengakumulasi mutasi lebih cepat daripada pseudogen yang tidak diproses.[21]
Referensi
- ^ "Pseudogen". www.institutoroche.es (dalam bahasa Spanyol). Diakses tanggal 22 November 2020.
- ^ Max EE (1986). "Plagiarized Errors and Molecular Genetics". Creation Evolution Journal. 6 (3): 34–46.
- ^ Chandrasekaran C, Betrán E (2008). "Origins of new genes and pseudogenes". Nature Education. 1 (1): 181.
- ^ Jurka J (December 2004). "Evolutionary impact of human Alu repetitive elements". Current Opinion in Genetics & Development. 14 (6): 603–8. doi:10.1016/j.gde.2004.08.008. PMID 15531153.
- ^ Dewannieux M, Heidmann T (2005). "LINEs, SINEs and processed pseudogenes: parasitic strategies for genome modeling". Cytogenetic and Genome Research. 110 (1–4): 35–48. doi:10.1159/000084936. PMID 16093656.
- ^ Dewannieux M, Esnault C, Heidmann T (September 2003). "LINE-mediated retrotransposition of marked Alu sequences". Nature Genetics. 35 (1): 41–8. doi:10.1038/ng1223. PMID 12897783.
- ^ Graur D, Shuali Y, Li WH (April 1989). "Deletions in processed pseudogenes accumulate faster in rodents than in humans". Journal of Molecular Evolution. 28 (4): 279–85. Bibcode:1989JMolE..28..279G. doi:10.1007/BF02103423. PMID 2499684.
- ^ Baertsch R, Diekhans M, Kent WJ, Haussler D, Brosius J (October 2008). "Retrocopy contributions to the evolution of the human genome". BMC Genomics. 9: 466. doi:10.1186/1471-2164-9-466. PMC 2584115
 . PMID 18842134.
. PMID 18842134.
- ^ Pavlícek A, Paces J, Zíka R, Hejnar J (October 2002). "Length distribution of long interspersed nucleotide elements (LINEs) and processed pseudogenes of human endogenous retroviruses: implications for retrotransposition and pseudogene detection". Gene. 300 (1–2): 189–94. doi:10.1016/S0378-1119(02)01047-8. PMID 12468100.
- ^ Navarro FC, Galante PA (July 2015). "A Genome-Wide Landscape of Retrocopies in Primate Genomes". Genome Biology and Evolution. 7 (8): 2265–75. doi:10.1093/gbe/evv142. PMC 4558860
 . PMID 26224704.
. PMID 26224704.
- ^ Schrider DR, Navarro FC, Galante PA, Parmigiani RB, Camargo AA, Hahn MW, de Souza SJ (2013-01-24). "Gene copy-number polymorphism caused by retrotransposition in humans". PLOS Genetics. 9 (1): e1003242. doi:10.1371/journal.pgen.1003242. PMC 3554589
 . PMID 23359205.
. PMID 23359205.
- ^ Max EE (2003-05-05). "Plagiarized Errors and Molecular Genetics". TalkOrigins Archive. Diakses tanggal 2020-11-23.
- ^ a b Lynch M, Conery JS (November 2000). "The evolutionary fate and consequences of duplicate genes". Science. 290 (5494): 1151–5. Bibcode:2000Sci...290.1151L. doi:10.1126/science.290.5494.1151. PMID 11073452.
- ^ Walsh JB (January 1995). "How often do duplicated genes evolve new functions?". Genetics. 139 (1): 421–8. PMC 1206338
 . PMID 7705642.
. PMID 7705642.
- ^ Lynch M, O'Hely M, Walsh B, Force A (December 2001). "The probability of preservation of a newly arisen gene duplicate". Genetics. 159 (4): 1789–804. PMC 1461922
 . PMID 11779815.
. PMID 11779815.
- ^ Harrison PM, Hegyi H, Balasubramanian S, Luscombe NM, Bertone P, Echols N, Johnson T, Gerstein M (February 2002). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Genome Research. 12 (2): 272–80. doi:10.1101/gr.207102. PMC 155275
 . PMID 11827946.
. PMID 11827946.
- ^ Zhang J (2003). "Evolution by gene duplication: an update". Trends in Ecology and Evolution. 18 (6): 292–298. doi:10.1016/S0169-5347(03)00033-8.
- ^ Nishikimi M, Kawai T, Yagi K (October 1992). "Guinea pigs possess a highly mutated gene for L-gulono-gamma-lactone oxidase, the key enzyme for L-ascorbic acid biosynthesis missing in this species". The Journal of Biological Chemistry. 267 (30): 21967–72. PMID 1400507.
- ^ Nishikimi M, Fukuyama R, Minoshima S, Shimizu N, Yagi K (May 1994). "Cloning and chromosomal mapping of the human nonfunctional gene for L-gulono-gamma-lactone oxidase, the enzyme for L-ascorbic acid biosynthesis missing in man". The Journal of Biological Chemistry. 269 (18): 13685–8. PMID 8175804.
- ^ Xue Y, Daly A, Yngvadottir B, Liu M, Coop G, Kim Y, Sabeti P, Chen Y, Stalker J, Huckle E, Burton J, Leonard S, Rogers J, Tyler-Smith C (April 2006). "Spread of an inactive form of caspase-12 in humans is due to recent positive selection". American Journal of Human Genetics. 78 (4): 659–70. doi:10.1086/503116. PMC 1424700
 . PMID 16532395.
. PMID 16532395.
- ^ Zheng D, Frankish A, Baertsch R, Kapranov P, Reymond A, Choo SW, Lu Y, Denoeud F, Antonarakis SE, Snyder M, Ruan Y, Wei CL, Gingeras TR, Guigó R, Harrow J, Gerstein MB (June 2007). "Pseudogenes in the ENCODE regions: consensus annotation, analysis of transcription, and evolution". Genome Research. 17 (6): 839–51. doi:10.1101/gr.5586307. PMC 1891343
 . PMID 17568002.
. PMID 17568002.
Bacaan selanjutnya
- Gerstein M, Zheng D (August 2006). "The real life of pseudogenes". Scientific American. 295 (2): 48–55. Bibcode:2006SciAm.295b..48G. doi:10.1038/scientificamerican0806-48. PMID 16866288.
- Torrents D, Suyama M, Zdobnov E, Bork P (December 2003). "A genome-wide survey of human pseudogenes". Genome Research. 13 (12): 2559–67. doi:10.1101/gr.1455503. PMC 403797
 . PMID 14656963.
. PMID 14656963. - Bischof JM, Chiang AP, Scheetz TE, Stone EM, Casavant TL, Sheffield VC, Braun TA (June 2006). "Genome-wide identification of pseudogenes capable of disease-causing gene conversion". Human Mutation. 27 (6): 545–52. doi:10.1002/humu.20335. PMID 16671097.
