Ekson
Ekson adalah bagian dari gen yang akan menyandi bagian dari RNA matang akhir yang dihasilkan oleh gen tersebut setelah intron telah dihilangkan oleh penjalinan RNA (RNA splicing). Istilah ekson mengacu pada urutan DNA dalam gen dan urutan yang sesuai dalam transkrip RNA. Dalam penjalinan RNA, intron dihapus dan ekson secara kovalen bergabung satu sama lain, menghasilkan RNA duta matang. Sama seperti seluruh rangkaian gen yang dimiliki suatu spesies membentuk genom, seluruh rangkaian ekson membentuk eksom.
Sejarah
Istilah ekson berasal dari wilayah yang diekspresikan dan dibuat oleh ahli biokimia Amerika Walter Gilbert pada tahun 1978: "Gagasan cistron ... harus diganti dengan unit transkripsi yang berisi daerah yang akan hilang dari duta matang – yang saya sarankan kita panggil intron (untuk wilayah intragenik) – bergantian dengan daerah yang akan diekspresikan – ekson. "[1]
Definisi ini awalnya dibuat untuk transkrip pengode protein yang dijalin sebelum ditranslasi. Istilah ini kemudian mencakup urutan yang dihapus dari rRNA[2] dan tRNA,[3] kemudian juga digunakan untuk molekul RNA yang berasal dari berbagai bagian genom yang kemudian disatukan dengan trans-splicing.[4]
Kontribusi untuk genom dan distribusi ukuran
Meskipun eukariota uniseluler seperti khamir tidak memiliki atau memiliki sangat sedikit intron, genom hewan dan terutama vertebrata memiliki sejumlah besar DNA non pengode. Misalnya, dalam genom manusia ekson hanya mencakup 1,1% dari genom, sedangkan 24% adalah intron, dan 75% genom merupakan DNA intergenik.[5] Ini dapat memberikan keuntungan praktis dalam perawatan kesehatan berbantuan omics (seperti pengobatan presisi) karena membuat pengurutan eksom keseluruhan yang dikomersialkan menjadi tantangan yang lebih kecil dan lebih murah daripada pengurutan genom keseluruhan yang dikomersialkan. Variasi besar dalam ukuran genom dan nilai C di antara makhluk hidup telah menimbulkan tantangan menarik yang disebut enigma nilai-C.
Dari semua gen eukariotik di GenBank, ada rata-rata 5,48 ekson per gen (pada tahun 2002). Setiap ekson rata-rata menyandikan 30-36 asam amino.[6] Sementara ekson terpanjang dalam genom manusia memiliki panjang 11555 bp, beberapa ekson memiliki panjang hanya 2 bp.[7] Ekson yang terdiri dari satu nukleotida telah dilaporkan dari genom Arabidopsis.[8]
Struktur dan fungsi
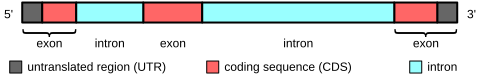
Dalam gen pengkode protein, ekson meliputi urutan pengkode protein dan daerah 5′ dan 3′ yang tidak ditranslasi (untranslated region, UTR). Seringkali ekson pertama mencakup 5′-UTR dan bagian pertama dari urutan pengode, tetapi ekson yang hanya berisi daerah 5′-UTR atau (lebih jarang) 3′-UTR terjadi pada beberapa gen, yaitu UTR dapat berisi intron.[9] Beberapa transkrip RNA non pengode juga memiliki ekson dan intron.
mRNA matang yang berasal dari gen yang sama tidak perlu menyertakan ekson yang sama, karena intron yang berbeda dalam pra-mRNA dapat dihilangkan dengan proses penjalinan alternatif.
Eksonisasi adalah penciptaan ekson baru, sebagai hasil mutasi pada intron.[10]
Pendekatan eksperimental menggunakan ekson
Penjebakan ekson (exon trapping) atau 'penjebakan gen' adalah teknik biologi molekuler yang mengeksploitasi keberadaan penjalinan intron-ekson untuk menemukan gen baru.[11] Ekson pertama dari gen yang 'terjebak' terjalin ke dalam ekson yang terkandung dalam DNA insersional. Ekson baru ini berisi ORF untuk gen reporter yang sekarang dapat diekspresikan menggunakan enhanser yang mengontrol gen target. Seorang ilmuwan tahu bahwa gen baru telah terperangkap ketika gen reporter diekspresikan.
Penjalinan dapat dimodifikasi secara eksperimental sehingga ekson yang ditargetkan dikeluarkan dari transkrip mRNA matang dengan memblokir akses partikel ribonukleoprotein nukleus kecil (small nuclear ribonucleoprotein particle, snRNP) yang mengatur penjalinan ke pra-mRNA menggunakan oligomer antisense Morpholino.[12] Ini telah menjadi teknik standar dalam biologi perkembangan. Oligomer Morpholino juga dapat ditargetkan untuk mencegah molekul yang mengatur penjalinan (mis. enhanser penjalinan, supresor penjalinan) untuk berikatan dengan pra-mRNA, sehingga mengubah pola penjalinan.
Lihat pula
- Exitron
- Pengocokan ekson
- Gen yang diinterupsi
- Intron
- mRNA
- Daerah yang tidak ditranslasi (untranslated region, UTR)
Referensi
- ^ Gilbert W (February 1978). "Why genes in pieces?". Nature. 271 (5645): 501. doi:10.1038/271501a0. PMID 622185.
- ^ Kister KP, Eckert WA (March 1987). "Characterization of an authentic intermediate in the self-splicing process of ribosomal precursor RNA in macronuclei of Tetrahymena thermophila". Nucleic Acids Research. 15 (5): 1905–20. doi:10.1093/nar/15.5.1905. PMC 340607
 . PMID 3645543.
. PMID 3645543.
- ^ Valenzuela P, Venegas A, Weinberg F, Bishop R, Rutter WJ (January 1978). "Structure of yeast phenylalanine-tRNA genes: an intervening DNA segment within the region coding for the tRNA". Proceedings of the National Academy of Sciences of the United States of America. 75 (1): 190–4. doi:10.1073/pnas.75.1.190. PMC 411211
 . PMID 343104.
. PMID 343104.
- ^ Liu AY, Van der Ploeg LH, Rijsewijk FA, Borst P (June 1983). "The transposition unit of variant surface glycoprotein gene 118 of Trypanosoma brucei. Presence of repeated elements at its border and absence of promoter-associated sequences". Journal of Molecular Biology. 167 (1): 57–75. doi:10.1016/S0022-2836(83)80034-5. PMID 6306255.
- ^ Venter J.C.; et al. (2000). "The Sequence of the Human Genome". Science. 291 (5507): 1304–51. doi:10.1126/science.1058040. PMID 11181995.
- ^ Sakharkar M, Passetti F, de Souza JE, Long M, de Souza SJ (2002). "ExInt: an Exon Intron Database". Nucleic Acids Res. 30 (1): 191–4. doi:10.1093/nar/30.1.191. PMC 99089
 . PMID 11752290.
. PMID 11752290.
- ^ Sakharkar M.K. et al (2004) Distributions of exons and introns in the human genome, In Silico Biology 4, 0032
- ^ Guo Lei, Liu Chun-Ming (2015). "A single-nucleotide exon found in Arabidopsis". Scientific Reports. 5: 18087. doi:10.1038/srep18087. PMC 4674806
 . PMID 26657562.
. PMID 26657562.
- ^ Bicknell, AA (December 2012). "Introns in UTRs: Why we should stop ignoring them". BioEssays. 34 (12): 1025–1034. doi:10.1002/bies.201200081. PMC 3710781
 . PMID 23065844.
. PMID 23065844.
- ^ Sorek R (October 2007). "The birth of new exons: mechanisms and evolutionary consequences". RNA. 13 (10): 1603–8. doi:10.1261/rna.682507. PMC 1986822
 . PMID 17709368.
. PMID 17709368.
- ^ Duyk G. M; Kim S. W.; Myers R. M; Cox D. R (1990). "Exon Trapping: a Genetic Screen to Identify Candidate Transcribed Sequences in Cloned Mammalian Genomic DNA". Proceedings of the National Academy of Sciences. 87 (22): 8995–8999. doi:10.1073/pnas.87.22.8995. PMC 55087
 . PMID 2247475.
. PMID 2247475.
- ^ Morcos PA (June 2007). "Achieving targeted and quantifiable alteration of mRNA splicing with Morpholino oligos". Biochemical and Biophysical Research Communications. 358 (2): 521–7. doi:10.1016/j.bbrc.2007.04.172. PMID 17493584.
Pustaka umum
- Zhang MQ (May 1998). "Statistical features of human exons and their flanking regions". Human Molecular Genetics. 7 (5): 919–32. doi:10.1093/hmg/7.5.919. PMID 9536098.
- Thanaraj TA, Robinson AJ (November 2000). "Prediction of exact boundaries of exons". Brief. Bioinform. 1 (4): 343–56. doi:10.1093/bib/1.4.343. PMID 11465052.
